DU GÉNOME AU PROTÉOME
Toutes les informations de l’organisme sont contenues dans notre ADN. Dans un gène, il y a plusieurs éléments : un site d’initiation de la transcription, une région promotrice, une séquence terminale dite de consensus, des exons et introns. Les exons constituent la région codant la protéine. Il existe des régions non codantes dans les introns et des séquences intergéniques qui régulent l’expression du génome. (1)
Pour que la séquence codante d’une protéine soit lue et exprimée, la double hélice d’ADN doit être exposée. Cela se produit grâce à l’acétylation des histones, responsables de la condensation de l’ADN.
De plus, une déméthylation de l’ADN et un signal de reconnaissance doivent se produire pour initier la transcription. L’ARN polymérase est activée, ce qui donnera naissance à un ARN messager. Par épissage, les introns sont séparés et les exons réunis entre eux et maintenus ainsi dans l’ARNm mature.
Le triplet de bases d’ARNm (codon) se lie à son complémentaire (anticodon) de l’ARN de transfert, puis se rendra au ribosome, où l’ARN ribosomal lira la séquence, commençant la traduction pour la synthèse de la protéine. Celle-ci sera nécessaire pour former une certaine structure ou intervenir dans une fonction organique. (2)

INTERFÉRENCE ARN
Après l’épissage, la plupart des séquences non codantes sont dégradées. Celles qui ne sont pas dégradées constituent les ARNs interférents (ARNi), régulateurs de l’expression génique.
Parmi les ARNs non codants, les miARNs (micro-ARNs interférents) ont une importance particulière. Leur biosynthèse se déroule comme suit :
1 ° Transcription du gène d’un miARN par l’ARN polymérase II: obtention d’une simple molécule d’ARN de mille nucléotides, le pri-mir ou miARN primaire.
Il contient une séquence non codante et une séquence complémentaire, ce qui permettra son appariement plus tard.
2 ° Splicing par la protéine Drosha: formation d’un ARN bicaténaire à 20 nucléotides, qui adopte une structure en “épingle à cheveux”, le pré-miR ou miARN précurseur.
3 ° Transport du pré-miR vers le cytoplasme cellulaire où, au moyen de la ribonucléase Dicer, est générée la séquence mature du miARN.
Le miARN est transporté vers le ribosome pour exercer son rôle modulateur dans la synthèse protéique, au moyen d’un appariement parfait avec l’ARNm cible.
Là, il module l’expression du gène codant à travers l’ensemble moléculaire RISC (RNA-induced silencing complex) par dégradation de l’ARNm ou blocage de sa traduction (le rendant illisible par les ribosomes) La synthèse de la protéine est empêchée. (3)
Les miARNs ont un rôle post-transcriptionnel clé. Mais ils peuvent aussi avoir un rôle pré-transcriptionnel, influençant les histones ou la méthylation de l’ADN. Ils agissent comme un mécanisme épigénétique de régulation génique. (4)
Ce mécanisme naturel des miARNs, l’interférence ARN, se produit en permanence dans nos cellules. Et il est de la plus haute importance qu’une unité transcriptionnelle soit exprimée ou que son expression soit réduite au silence.

HOMÉOSTASIE ET AUTO-RÉGULATION CELLULAIRE
L’ équilibre correct de notre système organique conduit au bon fonctionnement du corps. Des facteurs épigénétiques (environnement, nutrition, stress, miARNs, etc.) laissent des empreintes sur notre ADN, qui modulent l’expression des gènes.
Les miARNs régulent l’homéostasie cellulaire et participent à la différenciation, à la prolifération, à l’apoptose et au métabolisme des cellules. Ils sont également impliqués dans la réponse au stress, dans l’angiogenèse, l’oncogenèse et les processus cardiovasculaires. Mais ils peuvent également agir comme suppresseurs de tumeurs.
Il existe un grand nombre de miARNs chez l’humain
( actuellement on en connait déjà plus de 3000 ), qui régulent plus de 50% des gènes fonctionnels. L’existence de miARNs a également été retrouvée chez d’autres êtres vivants : animaux, plantes, champignons, etc. D’autres types d’ ARNs interférents sont également impliqués : siRNA (small interfering), piRNA (associé à piwi), etc.

LES MICROARNS EN TANT QUE BIOMARQUEURS
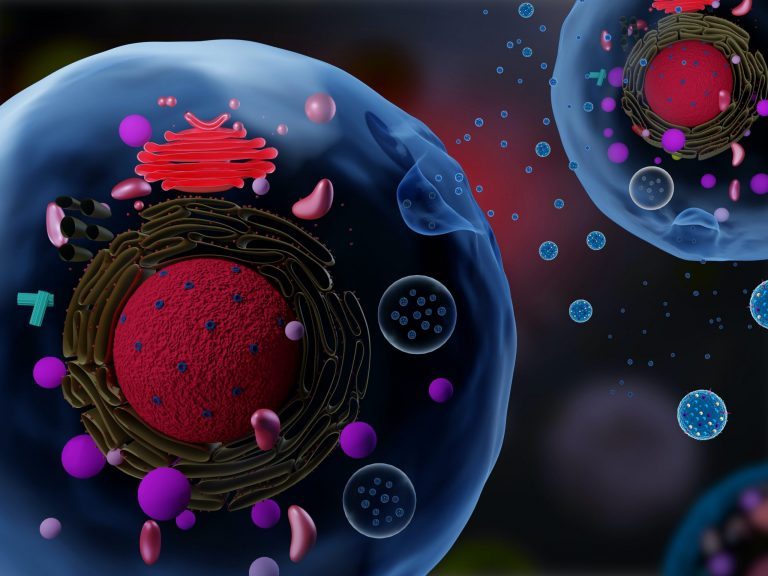
Les miARNs sont détectables dans les tissus et les fluides organiques (sérum, plasma, urine, salive, …) et abondants dans les exosomes et les microvésicules. (5)
Ils ont été liés à de nombreuses maladies: Parkinson, ostéoporose, cancer, diabète, hypertension, obésité, etc. Par conséquent, ils sont utiles comme biomarqueurs diagnostiques.
La dérégulation des miARNs peut conduire à une rupture de la tolérance immunitaire et, ensuite, au développement d’une maladie auto-immune. Elle peut aussi influencer la réponse immunitaire innée ou adaptative, en activant ou en arrêtant une infection. Une surexpression de miR-133a provoque une surexpression de RhoA dans les cellules musculaires lisses bronchiques, favorisant l’asthme allergique.
Les miARNs peuvent également servir de marqueurs pronostiques ou avancés de la maladie. Par exemple, miR-181a et miR-21 sont surexprimés dans l’ostéosarcome. Dans la forme plus agressive de cette maladie cancéreuse, il y a, en plus, une surexpression de miR-221-3p et une sous-expression de miR-222-3p, qu’on ne retrouve pas dans les formes à meilleur pronostic. MiR-199a est par ailleurs surexprimé dans l’ostéosarcome métastatique.
Il existe plusieurs bases de données publiées concernant les miARNs: Plos One, miRBase, mirtarbase, starbase, microRNA.org, etc.

BIO MÉDECINE IMMUNOGENETIQUE ET AUTO-RÉGULATION CELLULAIRE
Un avenir plein d’espoir s’ouvre pour le traitement des maladies, car on sait à l’heure actuelle, que les miARNs collaborent pour restaurer l’autorégulation cellulaire et l’homéostasie.
Le but de la Bio Médecine Immuno(G)énétique n’est pas l’introduction d’un médicament pour soigner un organe d’une manière ou d’une autre, mais d’aider la cellule à rétablir les voies et processus de signalisation modifiés dans le cadre de la maladie. Autrement dit, collaborer avec l’autorégulation cellulaire.
En utilisant une comparaison, si dans un orchestre il semble que la pièce musicale ne résonne pas comme elle le devrait, il vaut mieux d’abord savoir si le chef d’orchestre a commis une erreur dans l’interprétation du morceau ou s’il était distrait, avant de chercher à réparer ou de remplacer les instruments.
La Bio Médecine Immuno(G)énétique utilise des nanovecteurs pour introduire dans le corps les éléments naturels (puisqu’ils existent dans nos cellules) nécessaires pour restaurer les voies de signalisation altérées. Nous introduisons de la sorte des miARNs, ainsi que des protéines, des ligands, des enzymes, des hormones et des anticorps. Un seul miARN peut réguler de nombreux gènes cibles et influencer ainsi plusieurs voies de signalisation.
La BI(G)MED utilise des doses nanomoléculaires, c’est à dire des doses similaires à celles que l’on trouve à l’état normal dans nos cellules. Il s’agit d’apporter l’information sur les lieux d’action, d’augmenter l’efficacité et d’éviter les effets secondaires.
CONCLUSION
La découverte des ARN interférents, en particulier des miARNs, constitue une avancée très importante dans le diagnostic et le traitement des maladies, du fait de leur utilité comme biomarqueurs et comme outils thérapeutiques, sans les effets indésirables que les agents pharmacologiques traditionnels peuvent produire.
BIBLIOGRAPHIE
*https://en.wikipedia.org/wiki/MicroRNA
*https://www.exiqon.com/what-are-microRNAs
*Cavaillé, J. MicroRNA are everywhere. médecine/sciences. 2004. 20:p. 399-401
*Bernstein, E., Caudy, AA, et al. (2001) Role for a bidentate ribonuclease in the initiation step of RNA interference. Nature 409 (6818): 363-6
*Bartel, DP. MicroRNAs: genomics, biogenesis, merchanism, and function. Cell. 2004 Jan 23; 116 (2): 281-97.
*Zeng, Y., Cullen, BR. (2005) Efficient processing of primary microRNA hairpins by Drosha requires flanking nonstructured RNA sequences. J.Biol Chem. 2005. doi:10.1074/jbc.M504714200
*Kim, VN. MicroRNA biogenesis: coordinated cropping and dicing. Nat Rev Mol Cell Biol. 2005 May; 6 (5): 376-85
*Luc, C, Tej, et al. Elucidation of the small RNA component of the transcriptome. Science. 2005 Sep 2; 309(5740): 1567-9
*Pillai, RS. MicroRNA function: multiple mechanisms for a tiny RNA? RNA. 2005 Dec; 11(12):1753-1761.
*Lewis, BP. et al. Conserveds seed pairing, often flanked by adenosines indicates that thousands of human genes are microRNA targets. Cell 2005 Jan 14; 120 (1):15-20
*Hayley, B. and Zamore, P.D. Kinetic analysis of the RNA: enzyme complex. Nat. Struct Mol. Biol, 2004. 11(7): p 599-606
*Ameres, S.L.,Martínez, J., and Schroeder, R. Molecular basis for target RNA recognition and cleavage by human RISC. Cell. 2007, 130 (1):p 101-12
*Rico-Rosillo, MG., et al. Importancia de los microARN en el diagnóstico y desarrollo de
enfermedades. Rev. Med Inst Mex Seguro Soc. 2014; 52(3):302-7
*González Martín, A. microRNAs: pequeños reguladores con un gran impacto en nuestro sistema inmune. http://dx.doi.org/10.18567/sebbmdiv_RPC.2019.10.1
*Sasaki, R. et al. Micro RNA-Based Diagnosis and Treatment of Metastatic Human Osteosarcoma. Cancers (Basel) 2019 Apr; 11(4):553
*Jones, K., Salah, Z. et al. MicroRNA signatures associate with pathogenesis and progression of osteosarcoma. Cancer Res. 2012 Apr 1; 72(7):1865-1877
*Calin, GA. Et al. Frequent deletions and down-regulation of micro-RNA genes miR15 and miR16 at 13q14 in chronic lymphocytic leukemia. Proc Natl Acad sci USA. 2002 Nov 6;99(24):15524-9
FIGURES
1. Modification de l’histone (Shutterstock)
2. Synthèse des protéines par transcription (Shutterstock)
3. Biosynthèse des miARN (Wikimedia Commons)
4. Régulation pré et post-transcriptionnelle (Inmaculada Muñoz)
5. Exosomes détectables dans les fluides et utiles pour le diagnostic des maladies. Peut contenir des miARN, des protéines, de l’ARN, de l’ADN et des lipides de la cellule d’origine (Shutterstock)